Unidad Bioinformática y Biología Computacional
La unidad de Bioinformática y biología computacional (UBBC) proporciona servicios de análisis de datos bioinformáticos, ayudando a nuestros usuarios a comprender los resultados obtenidos a partir de grandes cantidades de datos, principalmente generados por experimentos de secuenciación masiva (NGS, next-generation sequencing technologies).
La UBBC es una unidad experta que ofrece soluciones bioinformáticas integrales en todos los aspectos de secuenciación y sus aplicaciones, utilizando algoritmos y pipelines de vanguardia, como así también en el análisis de imágenes de microscopia óptica y electrónica.

Servicios
Servicio Genómica y Transcriptómica
Análisis transcriptómico (RNA-seq)
Las tecnologías de secuenciación masiva (NGS) permiten el estudio de la expresión de los transcritos en diferentes procesos que ocurren en los organismos. De esta forma, se cuantifican simultáneamente miles de transcritos, generando millones de datos que dan cuenta de los diferentes procesos que ocurren en las células y/o tejidos.
Dependiendo del objetivo del análisis y de cómo se pre-traten las muestras previo a la secuenciación podemos agrupar nuestro análisis en las siguientes sub-áreas:
· Análisis de transcritos totales (Total RNA-seq)
· Análisis de transcritos mensajeros (mRNA-seq)
· Análisis de ARNs no codificantes pequeños (small RNA-seq)
· Análisis de ARNs no codificantes largos (long ncRNA)
Nuestros análisis se aplican en todo tipo de organismos (modelos y no modelos).
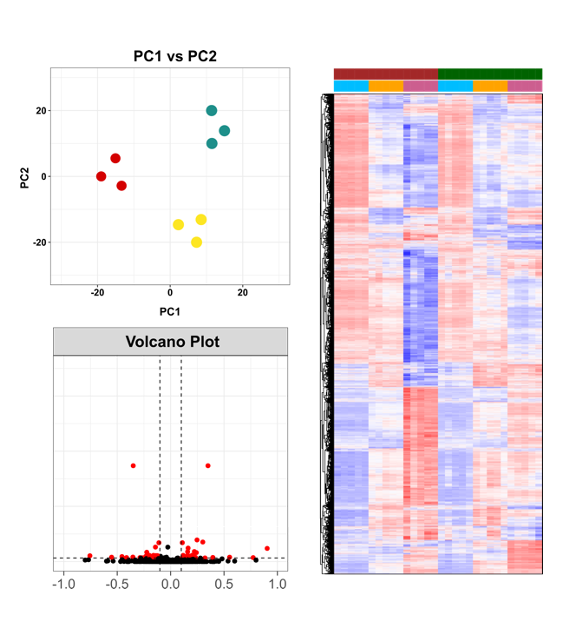
Por lo general, los usuarios están interesados en la expresión génica diferencial entre diferentes condiciones y/o a través del tiempo basada en secuenciación de ARN. En organismos modelos que cuentan con anotación esto considera:
- Demultiplexing
- Control de calidad de las lecturas obtenidas
- Filtrado y trimming de las lecturas
- Alineamiento de las lecturas contra una referencia (genoma/transcriptoma)
- Identificación y cuantificación de la expresión de los transcritos
- Análisis estadístico para la determinación de los genes/transcritos diferencialmente expresados
- Visualización de datos transcriptómicos
- Estudio de enriquecimiento de ontologías génicas y pathways en transcritos diferencialmente expresados
- Reconstrucción de novo del transcriptoma y su anotación
- Análisis de expresión de isoformas (splicing alternativo)
- Análisis de expresión allelo específico
- Identificación de fusión de genes
- Detección de variantes génicas (SNPs e indels)
- Análisis de co-expresión a partir de conjuntos de datos de expresión génica
Análisis genómicos
Las tecnologías de secuenciación permiten la determinación de la secuencia completa de ADN/ARN del genoma de un organismo. Con ello, se pueden caracterizar los genes presentes, identificar sus posibles funciones y encontrar variaciones genéticas (i.e. SNP).
En esta área la UBBC realiza los siguientes análisis:
- Secuenciación completa del genoma (de novo)
- Re-secuenciación del genoma
- Secuenciación de exomas (depende del organismo)
Nuestros análisis pueden ser personalizados de acuerdo a las necesidades del usuario.
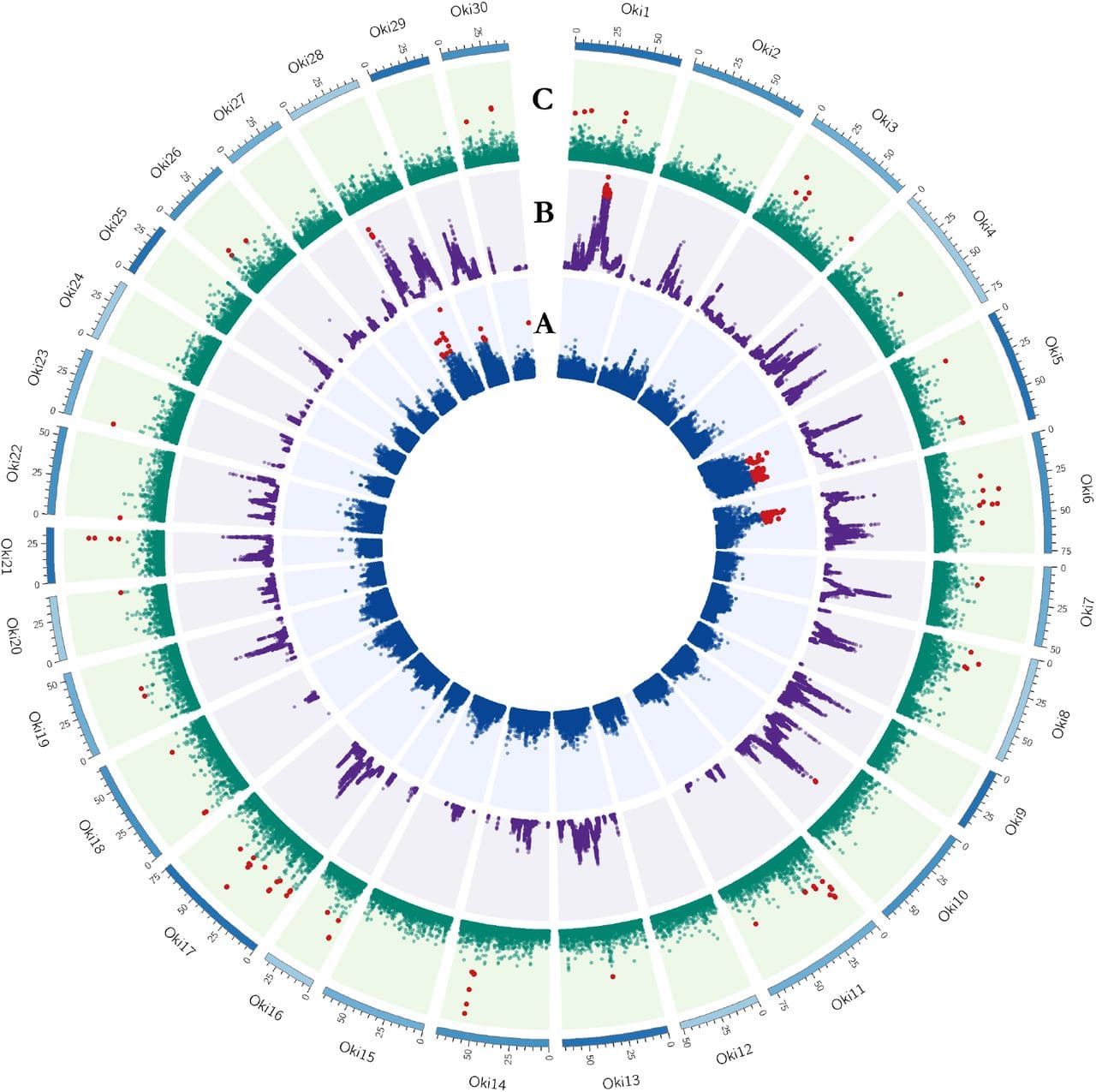
Aunque los análisis son diferentes, estos constan de una parte inicial similar que involucra por lo general lo siguiente
- Demultiplexing
- Control de calidad de las lecturas obtenidas
- Filtrado de las lecturas y trimming de las lecturas
- Detección de contaminantes
En particular, cada análisis considera lo siguiente:
Secuenciación completa del genoma (de novo)
- Ensamble de novo
- Refinamiento del ensamble
- Determinación taxonómica
- Predicción e identificación de genes
- Anotación funcional (ontologías génicas)
- Genómica comparativa (si el diseño experimental lo permite)
Re-Secuenciación del genoma
- Alineamiento de las lecturas a genoma de referencia
- Refinamiento del ensamble
- Detección de variaciones génicas (SNPs y Indels pequeños) y anotación
- Cálculo de métricas de cobertura y profundidad
- Genómica comparativa (si el diseño experimental lo permite)
- Determinación taxonómica
Secuenciación de exomas
- Alineamiento de las lecturas a exoma de referencia
- Detección de variaciones génicas (SNPs y Indels pequeños)
- Anotación de variaciones génicas (búsqueda en bases de datos)
- Comparación entre muestras
Análisis metagenómicos
La disciplina de la metagenómica permite estudiar la diversidad microbiana de nichos ecológicos. Ayuda a investigar la composición taxonómica en muestras como suelo, agua de mar, flora intestinal, flora respiratoria, etc.
En esta área la UBBC realiza los siguientes análisis:
- Análisis taxonómico mediante marcador 16S/18S/ITS (Metabarcoding)
- Análisis metagenómico shotgun
- Análisis de metatranscriptomica
- Identificación viral
Nuestros análisis pueden ser personalizados de acuerdo a las necesidades del usuario.
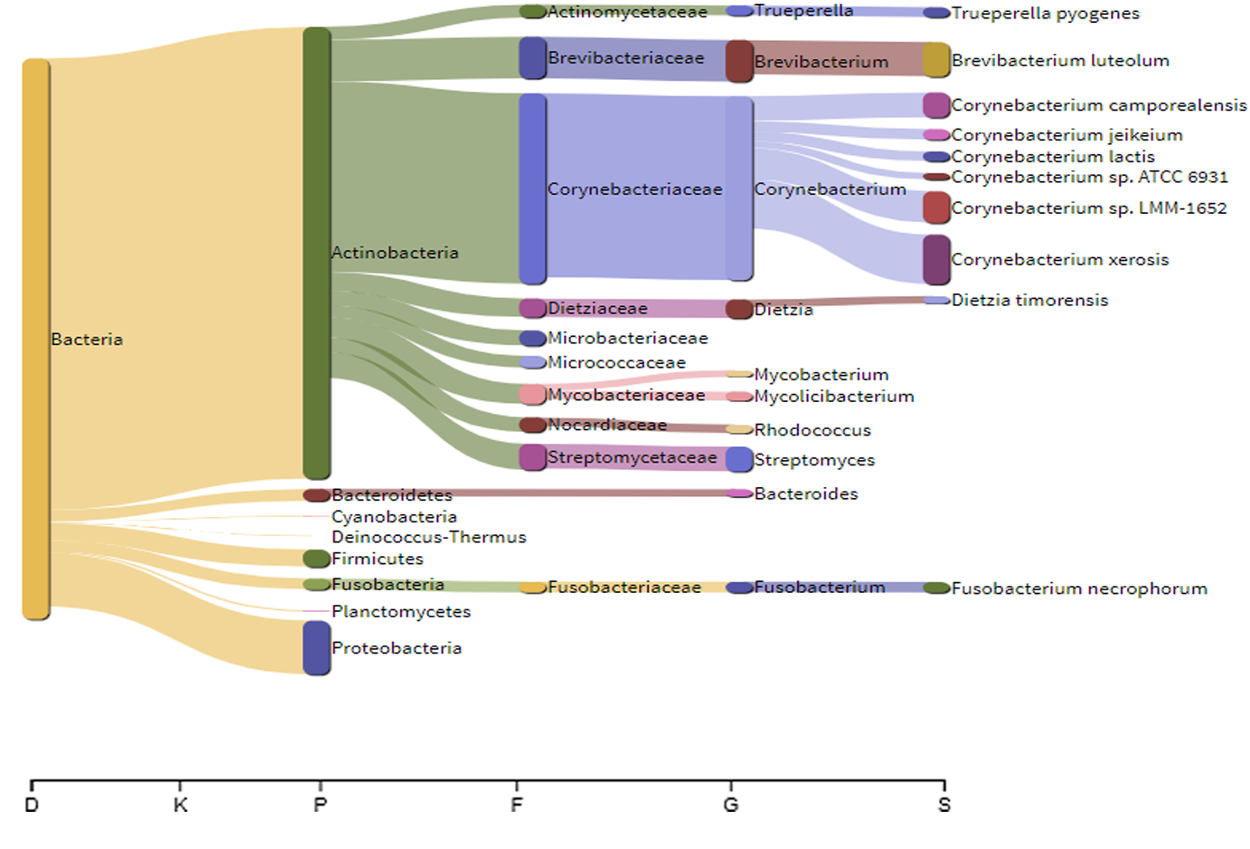
Aunque los análisis son diferentes, estos constan de una parte inicial similar que involucra por lo general lo siguiente:
- Demultiplexing
- Control de calidad de las lecturas obtenidas
- Filtrado de las lecturas (calidad, quimera, hospedero, ARN ribosomal y otros contaminantes)
En particular, cada análisis considera lo siguiente:
Análisis Taxonómico mediante marcador 16S/18S/ITS
- Obtención de tags. Unión de lecturas pair-end (depende de la metodología de secuenciación)
- Generación de ASVs (amplicon sequence variants)
- Filtro de ASVs
- Clasificación taxonómica de ASVs
- Cálculo de diversidad alfa
- Curvas de rarefacción
- Cálculo de diversidad beta
Análisis Metagenómico Shotgun
- Ensamble de contigs
- Refinamiento del ensamble
- Predicción e identificación de genes
- Anotación funcional (ontologías génicas)
- Anotación taxonómica
- Análisis de diversidad alfa
- Análisis de diversidad beta
- Análisis de rutas metabólicas
Análisis de Metatranscriptómica
- Reconstrucción de novo del transcriptoma
- Clustering de transcritos (transcritos NR)
- Anotación funcional de transcritos NR
- Cuantificación de la expresión de los transcritos
- Análisis de expresión diferencial
- Anotación taxonómica
- Análisis de enriquecimientos de ontologías
Análisis de Viromas (Identificación de virus)
- Unión de lexturas pair-end (depende de la metodología de secuenciación)
- Clustering de lecturas a un set no redundante
- Enmascaramiento de regiones repetitivas
- Descarte de lecturas que alinean con organismos no deseados
- Pipeline de busqueda de virus (i.e. VirusSeeker, ViromeScan u otro)